今天给大家介绍一个简单有效的网页工具,它可以帮助你绘制出精美的进化树圈图~~
iTOL网址:http://itol.embl.de/
首先先来看看用它绘制出来的漂亮图片吧
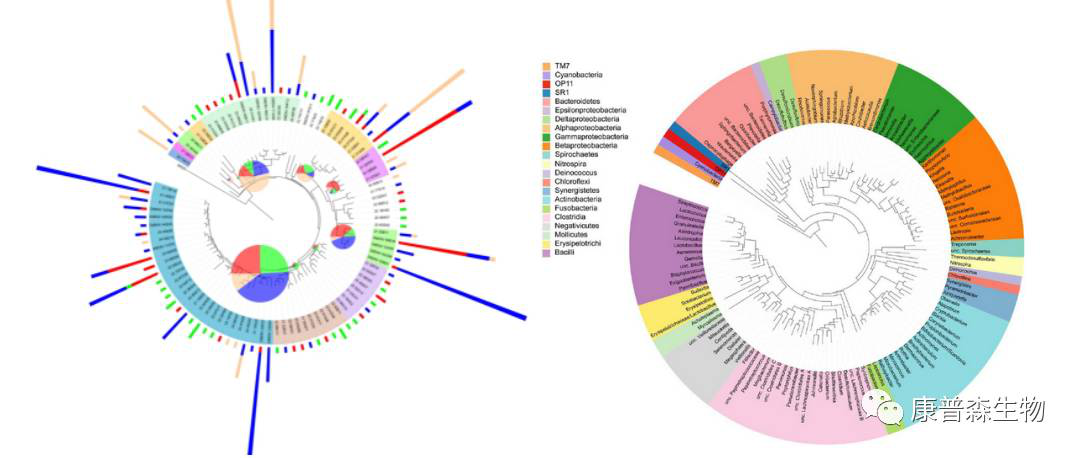
这两幅图片虽然看起来非常复杂,但是其实所需的数据类型很少,而且iTOL的操作界面也非常简洁。

网页主要分为5部分
1、图片展示区域。
2、图片缩放按钮:一般缩放图片可以通过鼠标滚轮进行操作,所以这两个按钮很少用到;此外,图片位置改变可以通过左键点击图片进行拖拽。
3、基本信息和搜索框:基本信息展示tree的名称和LEAF数目;搜索框可以通过LEAF名称在图片上凸显对应的位置。
4、control panel 为主要的控制面板,分为4个小panel:
Basic调节进化树的展示模式,Label的显示情况等;
Advanced调节进化树Bootstrap的展示情况等;
Datasets与第5部分的Colored ranges/datasets box配合使用;
Export控制图片输出属性;
5、Colored ranges/datasets box,这两个版面在一定的数据情况下才会显示,一般用来控制颜色和调节数据是否显示。
测试数据
如果你需要测试数据,你可以下载网址提供的example数据:
http://itol.embl.de/help/example_data.zip
操作很简单,在http://itol.embl.de/upload.cgi界面,将example数据中的tree_of_life.tree.txt文件拖拽上传即可。
上传完成后,点击文件名,即会跳转到iTOL图形操作界面,其它的文件可以直接拖拽到此界面即可以展示。
example 数据中文件主要分为以下三类:
1、进化树文件:tree_of_life.tree.txt,文件内容需要是Newick格式的。
2、进化树配置文件:用于配置进化树各node的颜色,label颜色等;数据会直接映射到进化树图形上,同时可以使用Colored ranges面板和Datasets面板调节。

3、Dataset types文件,用于添加到进化树圈外层的数据,同时可以使用datasets box面板和Datasets面板调节。
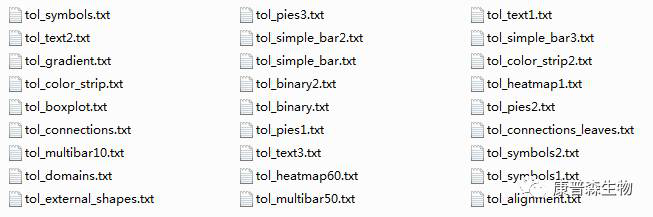
对于进化树配置文件和Dataset types文件,格式非常相似,其必须定义3个部分:
第一部分:声明需要的图形类型。
第二部分:声明文件中分隔符的类型。
第三部分:在DATA后指定实际的数据。
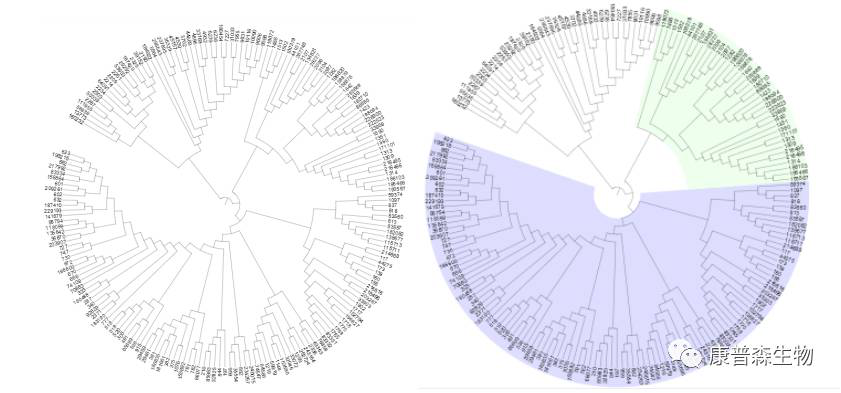
以文件中最简单的ranges.txt 为例:
数据的三个部分确定了数据是TREE_COLORS,用于给进化树上色的,分隔符是TAB键,分别要给I148和I100的分支区域上色。
将此文件拖拽到图形操作界面(操作界面首先要有tree_of_life.tree.txt的图形),tree即被上色:
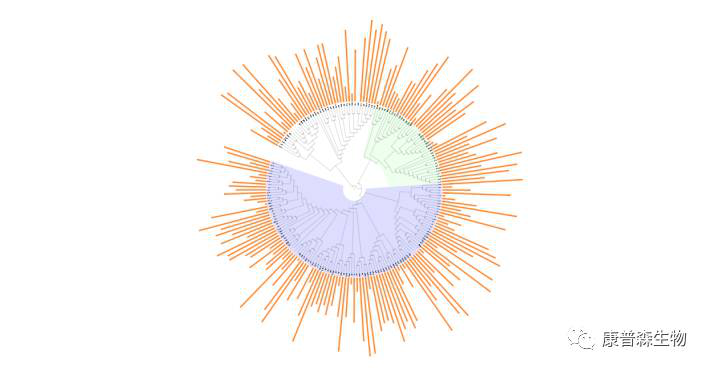
同样的,将tol_simple_bar2.txt文件拖拽到图形操作界面,即使用文件中的DATA数据,在进化树外圈展示数据:
您可以模仿example中的数据格式来配置自己的数据,即可得到相似的图形,非常方便。
当然,如果您还有不明白的话,iTOL网站也提供了非常友好的视频版的操作介绍:http://itol.embl.de/video_tutorial.cgi,视频时长约30min。通过视频学习,结合我们上面的简单讲解,相信您很快就能做出满意的图片了!也欢迎您随时与我们康普森生物公众号沟通!

 PREV
PREV
